复杂疾病中microRNA可以通过结合靶基因导致基因沉默,而RNA可以通过竞争性地结合microRNA来调节基因表达,这种调控称为ceRNA(competing endogenous RNAs,内源竞争RNA)。这揭示了一条RNA->microRNA调节通路的存在,对复杂疾病调控机制的研究具有重大生物意义。热带生物医学组学大数据李霞教授研究团队在《Briefings in Bioinformatics》杂志发表题为“CeRNASeek: an R package for identification and analysis of ceRNA regulation”文章(SCI 8.99,JCR一区)。
尽管目前有一些计算学方法被提出用于识别复杂疾病中ceRNA调控,然而这些方法散布于发表的文献中,目前没有有效的工具去系统识别复杂疾病中ceRNA的调控。为了解决这一难题,该研究开发了比较实用的工具CeRNASeek,集成于R语言环境中,整合了6种计算学方法用于识别复杂疾病中的ceRNA调控关系(图1)。用户只需要输入miRNA-gene的调控或者miRNA/gene的表达数据,利用该工具可以识别ceRNA调控网络,并对网络和ceRNA的功能进行分析,还能有效地识别复杂疾病中与预后相关的ceRNA调控模块。
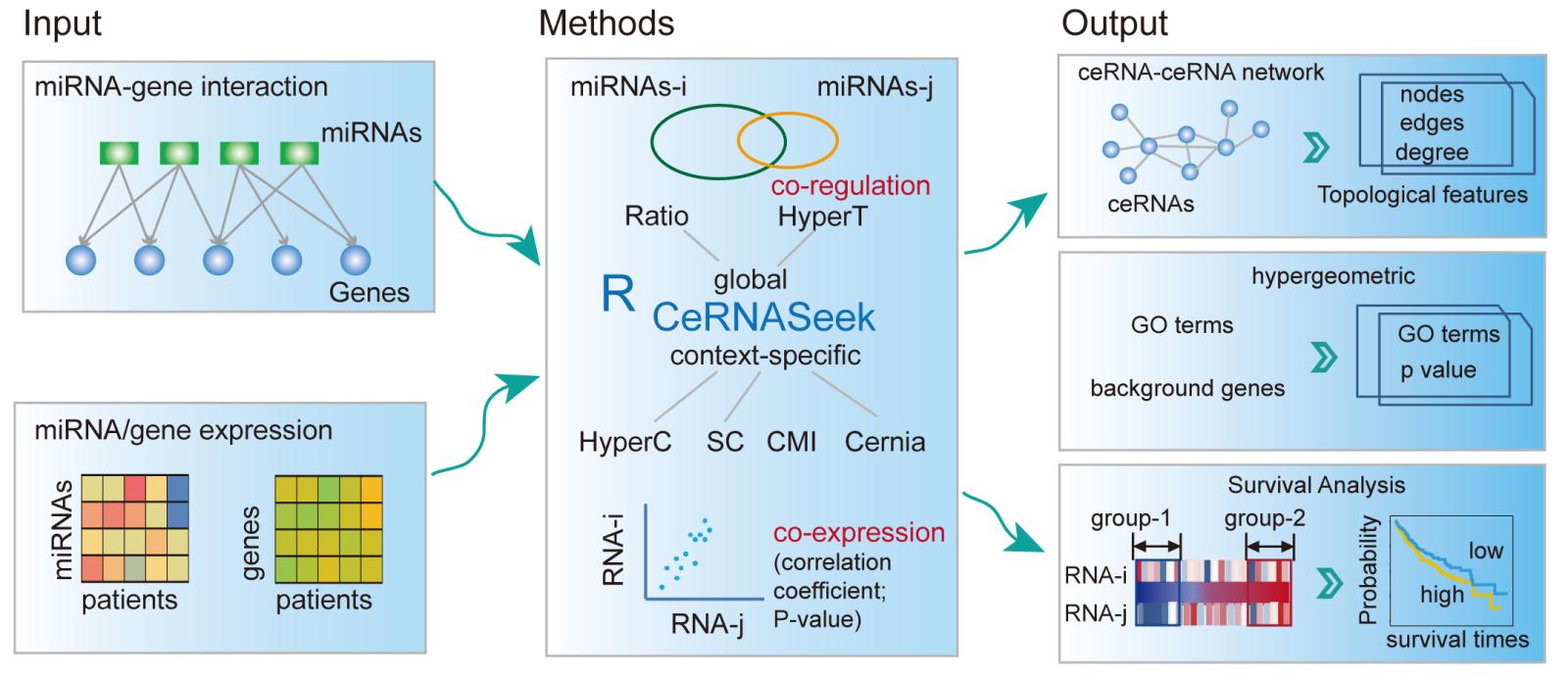
图1、CeRNASeek工具构建流程
以PTEN相关的ceRNA调控为研究案例,该研究系统识别了多种癌症中PTEN参与的ceRNA调控,并构建了调控网络(图2)。比较不同方法发现,这些计算方法间是互补的,该工具的建立能够快速的整合不同的计算方法,有效地识别ceRNA调控,并进行功能分析。功能富集分析发现这些PTEN相关的ceRNA基因广泛地参与癌症相关的生物学通路。此外,将该工具应用于泛癌的研究,识别了癌症中的ceRNA调控网络,特别地,在基于ceRNA调控识别了肝癌的不同分子亚型,这些不同亚型的病人间在突变、生存以及治疗反应中具有显著的差异。这些结果充分证实了该工具的实用性。
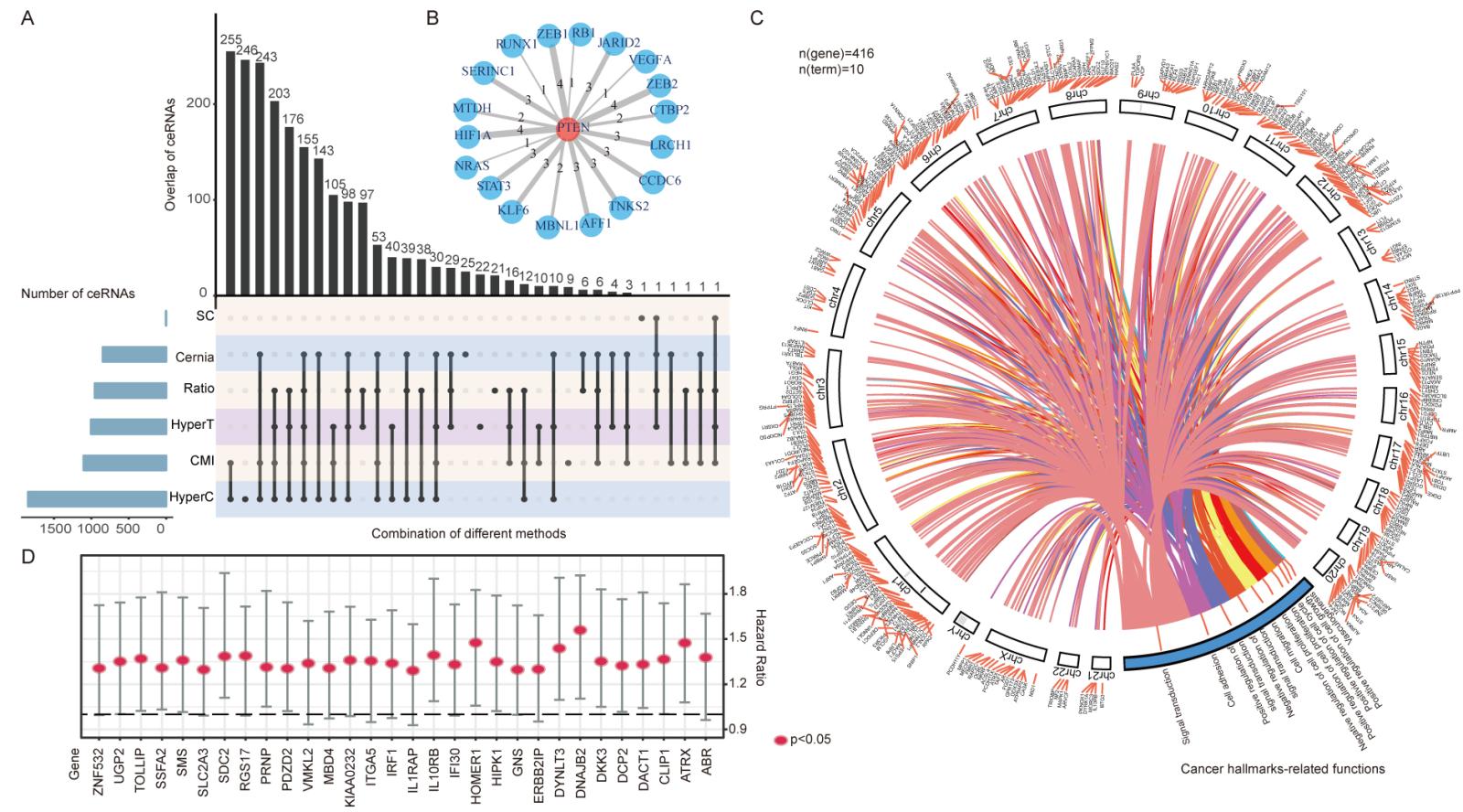
图2、PTEN相关ceRNA调控网络。
李霞教授研究团队一直关注于复杂疾病中ceRNA的作用机制研究,开发了一系列计算学方法解析ceRNA在疾病中的调控作用方式。这些研究结果发表于Nucleic Acids Research、Briefings in Bioinformatics等国际著名杂志。该项研究工作受到了国家自然科学基金(61873075, 31871338, 31970646)以及热带转化医学教育部重点实验室等项目的资助。
全文链接:https://pubmed.ncbi.nlm.nih.gov/32363380/